Construção arranjando em sequência eficaz na redução de custos Kit From Chinese Manufacturers da biblioteca
Detalhes do produto:
| Lugar de origem: | China |
| Marca: | GDSBio |
| Certificação: | ISO9001, ISO13485 |
| Número do modelo: | KM001-A, KM001-B |
Condições de Pagamento e Envio:
| Quantidade de ordem mínima: | 1 jogo |
|---|---|
| Preço: | US $0.2-0.75 / Piece |
| Detalhes da embalagem: | Encaderne o empacotamento |
| Tempo de entrega: | dias 8work |
| Termos de pagamento: | L/C, D/A, D/P, T/T, Western Union, MoneyGram |
| Habilidade da fonte: | 5000 jogos/semana |
|
Informação detalhada |
|||
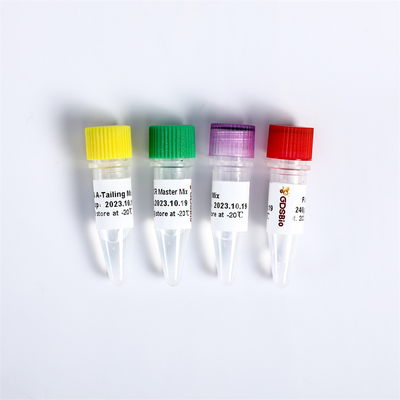
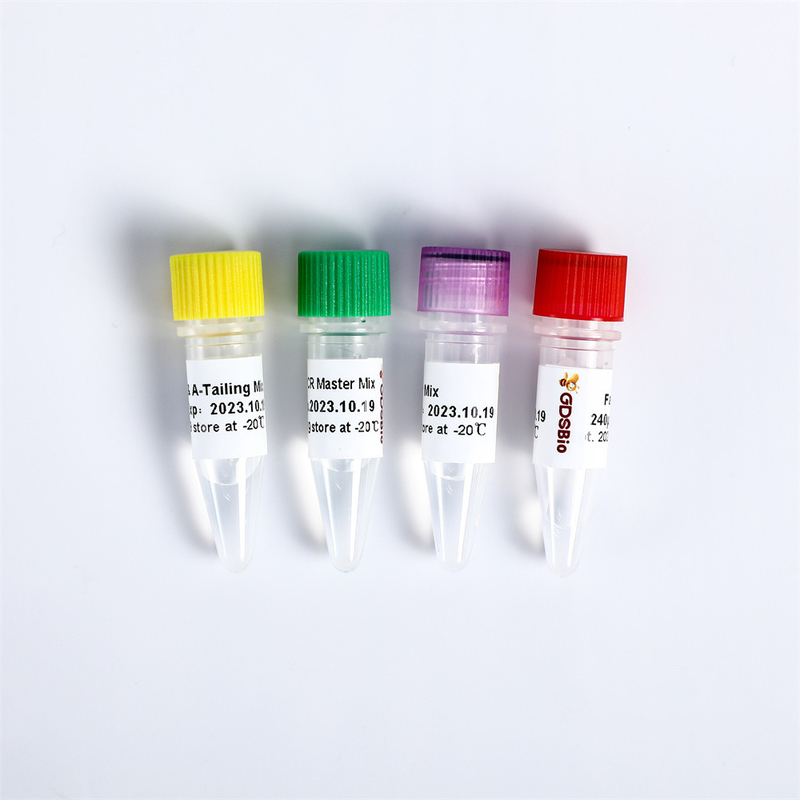
| Gato. Não.: | KM001-A, KM001-B | Especificação do tubo: | Rxns KM001-A/24; Rxns KM001-B/96 |
|---|---|---|---|
| Arranjando em sequência a plataforma: | MGI | Material entrado: | ADN |
| Vida útil: | 12 meses | Armazenamento: | -20°C |
| Realçar: | Tubo de amostra descartável do vírus da inativação,Tubo de amostra descartável do vírus de FDA,VTM neutralizou o tubo de amostra do vírus |
||
Descrição de produto
Jogo rápido da preparação da biblioteca do ADN para MGI
[Nome do produto]
Jogo rápido da preparação da biblioteca do ADN para MGI
[Gato. Não/especs.]
Rxns KM001-A/24; Rxns KM001-B/96; rxns do saco 6 da amostra
[Descrição do produto]
Visando a alto-taxa de transferência de MGI que arranja em sequência a plataforma, este jogo fornece um esquema de construção conveniente e universal da biblioteca do ADN em um tubo. Combina o reparo e a Um-pedra saliente da extremidade em uma etapa, e os reagentes premixed altamente, encurtando extremamente a época da construção da biblioteca e reduzindo o erro causado por etapas fastidiosas. A preparação da extremidade, a ligadura do adaptador, a amplificação e a purificação do ADN dobro-encalhado fragmentado podem ser executadas dentro de cerca de 2 horas. A quantificação completa da biblioteca pode ser executada pelo método da tintura fluorescente do dsDNA (por exemplo, Qubit Thermo Flex Fluorometer) ou pelo PCR absoluto da quantificação após ter diluído a biblioteca a uma concentração apropriada.
[Tipo da amostra]
| Aplicação | Tipo da amostra | Uma quantidade recomendada |
| Arranjar em sequência inteiro do genoma | Genomas complexos de alta qualidade | 50ng-1μg |
| Arranjar em sequência da captação do alvo do exome inteiro | Genomas complexos de alta qualidade | 10ng-1μg |
| Arranjar em sequência da captação do alvo do genoma inteiro | ADN DE FFPE | ≥50ng |
| Arranjar em sequência da captação do alvo do genoma inteiro | cfDNA/ctDNA | ≥100pg |
| Arranjar em sequência inteiro do genoma | Genoma microbiano | 1ng-1μg |
| Microplaqueta-segs. | ADN da microplaqueta | ≥100pg |
| Arranjar em sequência visado | Amplicon | ≥100pg |
[Condição de armazenamento & vida útil]
Todos os reagentes devem ser armazenados no amortecedor da ligadura de -20°C. são normais para que os cristais precipitem em baixas temperaturas, ele devem ser equilibrados à temperatura ambiente antes de usar. O produto é válido por 12 meses.
[Componentes]
| Componente | 24 rxns | 96 rxns |
| Reparo do fim & mistura da Um-pedra saliente | μl 480 | μl 4×480 |
| Ligase rápida do ADN | μl 120 | μl 2×240 |
| Amortecedor rápido da ligadura | μl 600 | μl 4×600 |
| Mistura mestra do PCR da biblioteca 2× DE ALTA FIDELIDADE | μl 600 | μl 4×600 |
| Mistura da primeira demão para MGI* | μl 120 | μl 480 |
* se há mais de uma amostra, a mistura da primeira demão do adaptador #KM002 e #KM003 está recomendada. Este jogo fornece um grupo de primeiras demão.
Nota: grânulos recomendados da seleção: Grânulos da seleção Magbeads ou do AMPure XP do ADN de #NC1011 GDSPure.
[Notas]
1. Nós oferecemos dois tipos de grupo universal das primeiras demão do adaptador (#KM002 e #KM003, comprados separadamente), mas os clientes podem igualmente escolher de outros fabricantes ou sintetizar seu próprio adaptador para o MGI que arranja em sequência a plataforma. Demasiado adaptador conduzirá à formação de dímero do adaptador, e o insuficiente adaptador conduzirá à baixa saída da biblioteca. Consequentemente, a concentração apropriada do adaptador determina a concentração e a qualidade da biblioteca. As concentrações recomendadas do adaptador para quantidades diferentes de entrada do ADN são mostradas na seguinte tabela:
Concentrações recomendadas do uso da tabela 1 de adaptador
| O ADN entrou | Concentrado recomendado para o adaptador | Adaptador: Relação de toupeira da inserção | Diluição Degrees* do adaptador de GDS |
| 1μg | 10μM | 10:1 | Nenhuma diluição |
| 500ng | 10μM | 20:1 | Nenhuma diluição |
| 250ng | 10μM | 40:1 | Nenhuma diluição |
| 100ng | 7.5μM | 100:1 | 3:4 |
| 50ng | 5μM | 200:1 | 1:2 |
| 25ng | 2.5μM | 200:1 | 1:4 |
| 1ng | 1μM | 200:1 | 1:10 |
* expressado como a relação do volume do adaptador ao diluente
2. A enzima usada na mistura mestra do PCR da biblioteca 2× DE ALTA FIDELIDADE é uma polimerase de ADN da família de B, que tenha 5" - 3" polimerase e 3" - 5" atividades do exonuclease, mas falta 5" - 3" atividades do exonuclease. Tem a alta fidelidade e a homogeneidade, e a capacidade sustentável forte da síntese. O controle restrito do número de ciclos da amplificação é particularmente importante para a saída da biblioteca. A seguinte tabela mostra o número recomendado de ciclos da amplificação que correspondem às quantidades diferentes de entrada do ADN:
Número recomendado da tabela 2 de ciclos da amplificação que correspondem às entradas diferentes da amostra
| ADN entrado | Número recomendado de ciclos da amplificação | |
| biblioteca 100ng | biblioteca 1μg | |
| 1μg | 0 | 2-5 |
| 500ng | 0 | 2-5 |
| 250ng | 1-3 | 5-7 |
| 100ng | 2-4 | 6-8 |
| 50ng | 4-6 | 8-10 |
| 25ng | 5-7 | 9-12 |
| 10ng | 7-9 | 11-13 |
| 5ng | 9-11 | 13-14 |
| 2.5ng | 10-12 | 14-16 |
| 1ng | 11-13 | 15-17 |
Nota: 1. A tabela acima mostra os resultados da análise usando 150bp o ADN padrão, que é para a referência somente.
2. Se os conectores incompletos são usados, um número mínimo dos ciclos (1-3) deve ser amplificado para obter uma biblioteca completa.
3. Se a qualidade do ADN da entrada é pobre, ou a seleção de tamanho está realizada durante a construção da biblioteca, o número de ciclos da amplificação deve apropriadamente ser aumentado.
[Processo padrão da construção da biblioteca]
Reparo do fim
Nota: Se o ADN fragmentado excede o μl 45 antes que esta etapa, ou o amortecedor estejam incompatíveis com o amortecedor do reparo do fim, uma purificação magnética do grânulo deve ser executada primeiramente.
1. Prepare a seguinte reação em um tubo do PCR de 200 μl:
| Reagentes | Volume |
| ADN fragmentado | Variável |
| Reparo do fim & mistura da Um-pedra saliente | μl 20 |
| ddH2 O | Ao μl 65 |
2. Redemoinho delicadamente e rotação para baixo momentaneamente a misturar bem, centrifugar momentaneamente e recolher todo o líquido à parte inferior do tubo.
3. Execute a seguinte reação em um cycler térmico:
| Temperatura | Tempo |
| 20°C | 15min |
| 65°C | 15min |
| 4°C | ∞ |
Ligadura do adaptador
1. Continue com a reação da ligadura o mais cedo possível após a preparação da extremidade.
2. Dilua o adaptador de acordo com a tabela 1.
3. Prepare o seguinte sistema de reação:
| Reagentes | Volume |
| Reparo do fim e produtos da Um-pedra saliente | μl 65 |
| Amortecedor rápido da ligadura | μl 25 |
| Ligase rápida do ADN | μl 5 |
| Adaptador X para MGI | μl 5 |
| Total | μl 100 |
4. Redemoinho delicadamente e rotação para baixo momentaneamente a misturar bem, centrifugar momentaneamente e recolher todo o líquido à parte inferior do tubo.
5. Execute a seguinte reação em um cycler térmico:
| Temperatura | Tempo |
| 20°C | 15min |
| 4°C | ∞ |
Solução recomendada para a limpeza do PCR/seleção de tamanho (o volume magnético específico do grânulo deve ser ajustado de acordo com o tamanho da amostra real)
1. Prepare 100 produtos da ligadura do μl em um tubo de centrifugador apropriado.
2. Adicione o μl 100 de grânulos magnéticos resuspended da seleção do ADN à amostra. Delicadamente sopro com uma pipeta por 10 vezes (ou redemoinho para 30 s). Incube amostras para o minuto 5 na temperatura ambiente.
3. Coloque o tubo em uma cremalheira magnética apropriada para separar os grânulos do supernatant. Quando a solução for clara, para remover e rejeitar com cuidado o supernatant com uma pipeta (não rejeite grânulos).
4. Adicione o μl 200 do álcool etílico recentemente preparado de 80% ao tubo quando na cremalheira magnética. Incube na temperatura ambiente para 30 s, e para remover então e rejeitar com cuidado o supernatant (não perturbe grânulos).
5. Repita etapa 4 uma vez para um total de duas lavagens.
Nota: Seja certo remover todo o líquido visível após o segundo Washington.
6. O ar seca os grânulos até que a superfície dos grânulos magnéticos não tenha nenhum brilho óbvio quando o tubo estiver na cremalheira magnética com a tampa aberta.
Nota: Faça não overdry os grânulos, este pode conduzir a uma mais baixa recuperação do ADN. Quando os grânulos começam se rachar, estão demasiado secos.
7. Remova o tubo da cremalheira magnética. Adicione o amortecedor da eluição de 22 μl (Tris-HCl, pH8.0-8.5 de 10mM) ao tubo. Misture bem introduzindo com pipeta para cima e para baixo pelo menos 10 vezes ou em um misturador do redemoinho para 30 o S. incuba para o minuto 3-5 na temperatura ambiente.
8. Coloque o tubo na cremalheira magnética. Após o minuto 5 (ou quando a solução for clara), supernatant do μl de transferência 20 a um tubo novo. A seleção é terminada, e o ADN selecionado pode ser usado para experiências subsequentes ou ser armazenado em -20°C por muito tempo.
Amplificação da biblioteca
1. Prepare a seguinte reação em um tubo do PCR de 200 μl:
| Reagentes | Volume |
| Produtos da ligadura após a seleção da limpeza ou de tamanho | μl 20 |
| Mistura mestra do PCR da biblioteca 2× DE ALTA FIDELIDADE | μl 25 |
| Mistura da primeira demão para MGI | μl 5 |
| Total | μl 50 |
2. Redemoinho delicadamente e rotação para baixo momentaneamente a misturar bem, centrifugar momentaneamente e recolher todo o líquido à parte inferior do tubo.
3. Execute a seguinte reação em um cycler térmico:
| Temperatura | Tempo | Número de ciclo |
| 95°C | 3min | 1 |
| 98°C | 20sec |
Número apropriado seleto de ciclos de acordo com a tabela 2 |
| 60°C | 15sec | |
| 72°C | 30sec | |
| 72°C | 5min | 1 |
| 4°C | ∞ | - |
Solução recomendada para a limpeza do PCR/seleção de tamanho (o volume magnético específico do grânulo deve ser ajustado de acordo com o tamanho da amostra real)
1. Prepare 50 produtos da ligadura do μl em um tubo de centrifugador apropriado.
2. Adicione o μl 45 de grânulos magnéticos resuspended da seleção do ADN à amostra. Delicadamente sopro com uma pipeta por 10 vezes (ou redemoinho para 30 s). Incube amostras para o minuto 5 na temperatura ambiente.
3. Coloque o tubo em uma cremalheira magnética apropriada para separar os grânulos do supernatant. Quando a solução for clara, para remover e rejeitar com cuidado o supernatant com uma pipeta (não rejeite grânulos).
4. Adicione o μl 200 do álcool etílico recentemente preparado de 80% ao tubo quando na cremalheira magnética. Incube na temperatura ambiente para 30 s, e para remover então e rejeitar com cuidado o supernatant (não perturbe grânulos).
5. Repita etapa 4 uma vez para um total de duas lavagens.
Nota: Seja certo remover todo o líquido visível após o segundo Washington.
6. O ar seca os grânulos até que a superfície dos grânulos magnéticos não tenha nenhum brilho óbvio quando o tubo estiver na cremalheira magnética com a tampa aberta.
Nota: Faça não overdry os grânulos, este pode conduzir a uma mais baixa recuperação do ADN. Quando os grânulos começam se rachar, estão demasiado secos.
7. Remova o tubo da cremalheira magnética. Adicione o amortecedor da eluição de 22 μl (Tris-HCl, pH8.0-8.5 de 10mM) ao tubo. Misture bem introduzindo com pipeta para cima e para baixo pelo menos 10 vezes ou em um misturador do redemoinho para 30 o S. incuba para o minuto 3-5 na temperatura ambiente.
8. Coloque o tubo na cremalheira magnética. Após o minuto 5 (ou quando a solução for clara), supernatant do μl de transferência 20 a um tubo novo. A seleção é terminada, e o ADN selecionado pode ser armazenado em 2-8°C por 1-2 semanas ou ser armazenado em -20°C por muito tempo.
[Apêndice] esquema recomendado para seleção frente e verso
Se a seleção dobro-redonda é exigida, nós fornecemos o seguinte esquema para selecionar o volume magnético apropriado do grânulo de acordo com o tamanho previsto da biblioteca. A seleção de tamanho pode ser executada antes do reparo do fim ou após a amplificação. Uma seleção dois ou dobro-mais redondos reduzirá extremamente o rendimento da biblioteca.
Encha o volume da biblioteca na tabela abaixo ao μl 100. Selecione o volume de grânulos magnéticos em dois círculos de acordo com o tamanho previsto da biblioteca. E realize a operação da seleção de acordo com as seguintes instruções.
Uma quantidade recomendada da tabela 3 de grânulos magnéticos para a seleção Dobro-redonda
| Tamanho previsto da biblioteca | 150bp | 200bp | 250bp | 300bp | 400bp | 500bp | 600bp | 700bp | |
| Volume de grânulos (μl) | Círculo 1 | 100 | 90 | 80 | 70 | 60 | 55 | 50 | 45 |
| Círculo 2 | 30 | 20 | 20 | 20 | 20 | 15 | 15 | 15 | |
1. Encha o volume da biblioteca ao μl 100 em um tubo do PCR 200μl e etiquetado como A. Adição algum volume de grânulos magnéticos de acordo com a tabela 3 (círculo 1) ao sopro de A. Delicado do tubo com uma pipeta para 30 S. incuba amostras para o minuto 5 na temperatura ambiente.
2. Coloque o tubo A em uma cremalheira magnética apropriada para separar os grânulos do supernatant. Quando a solução é clara, remova com cuidado o supernatant a um tubo novo e etiquete-o como grânulos de B. Rejeição.
3. Adicione algum volume de grânulos magnéticos de acordo com a tabela 3 (círculo 2) ao sopro de B. Delicado do tubo com uma pipeta para 30 que o S. incuba amostras para o minuto 5 na temperatura ambiente. Coloque o tubo B na cremalheira magnética. Quando a solução for clara, para remover e rejeitar com cuidado o supernatant.
4. Adicione o μl 200 do álcool etílico recentemente preparado de 80% ao tubo B quando na cremalheira magnética. Incube na temperatura ambiente para 30 s, e para remover então e rejeitar com cuidado o supernatant (não perturbe grânulos).
5. Repita etapa 6 uma vez para um total de duas lavagens.
Nota: Seja certo remover todo o líquido visível após o segundo Washington.
6. O ar seca os grânulos até que a superfície dos grânulos magnéticos não tenha nenhum brilho óbvio quando o tubo B estiver na cremalheira magnética com a tampa aberta.
Nota: Faça não overdry os grânulos, este pode conduzir a uma mais baixa recuperação do ADN. Quando os grânulos começam se rachar, estão demasiado secos.
7. Remova o tubo B da cremalheira magnética. Adicione o amortecedor da eluição de 22 μl ao tubo. Misture bem introduzindo com pipeta para cima e para baixo pelo menos 10 vezes ou em um misturador do redemoinho para 30 o S. incuba para o minuto 3-5 na temperatura ambiente.
Nota: Se a captação visada não será executada, adicione o amortecedor da eluição (Tris-HCl de 10mM, pH 8.0-8.5) para a eluição. Se não, a água ultrapure esterilizada deve ser usada para a eluição.
- Tubo B do lugar na cremalheira magnética. Supernatant do μl de transferência 20 a um tubo novo.
Para o uso da pesquisa somente
GDSBio é uma empresa da alto-tecnologia que centra-se sobre a investigação e desenvolvimento, a produção e as vendas dos produtos de alta qualidade da ciência da vida. A empresa tem uma linha de produtos completa, com a tecnologia do PCR como o núcleo, centrando-se sobre o PCR geral, armazenamento quantitativo do PCR da fluorescência, do NGS, eletroforese ácida nucleica e outras tecnologias da biologia molecular, e desenvolveu reagentes moleculars da pesquisa, in vitro matérias primas diagnósticas moleculars, reagentes da extração ácida nucleica e da detecção e outros produtos.